yolo를 배웠기때문에 YOLO로 폐질환 환자를 구별해보려고 합니다.
KAGGEL데이터를 가져올것이기때문에 KAGGEL을 가지고 옵니다.(API)
#캐글 데이터셋 다운로드
!kaggle datasets download -d hamdallak/the-iqothnccd-lung-cancer-dataset# 압축 풀기
!unzip -q /content/the-iqothnccd-lung-cancer-dataset.zip압축을 풀게되먄 The IQ-OTHNCCD lung cancer dataset 가 생성됨
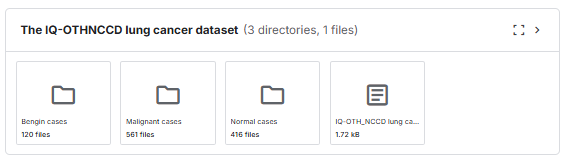
[ Begin - 시작되는단계, Malignat - 폐암, Normal - 일반인 ]
이렇게 시작, 폐암, 일반인으로 3가지의 분류가 되어있는것을 확인할 수 있습니다.
YOLO는 울트라틱스에있기때문에 기본적으로 설치를 해야합니다.
!pip install ultralytics🗒️ 필요한 모듈 임포트
#필요한 모듈 임포트
import os
import random
import shutil
import cv2
import glob
import yaml
import matplotlib.pyplot as plt
import ultralytics
import numpy as np
import torch
from torchvision import transforms
from tqdm import tqdm
from PIL import Image
from ultralytics import YOLO
필요한 모듈 모두 위쪽에 적어두기로 했습니다.
그리고 random.seed(2024)로 잡아주었습니다.
🗒️ 데이터 전처리 준비하기
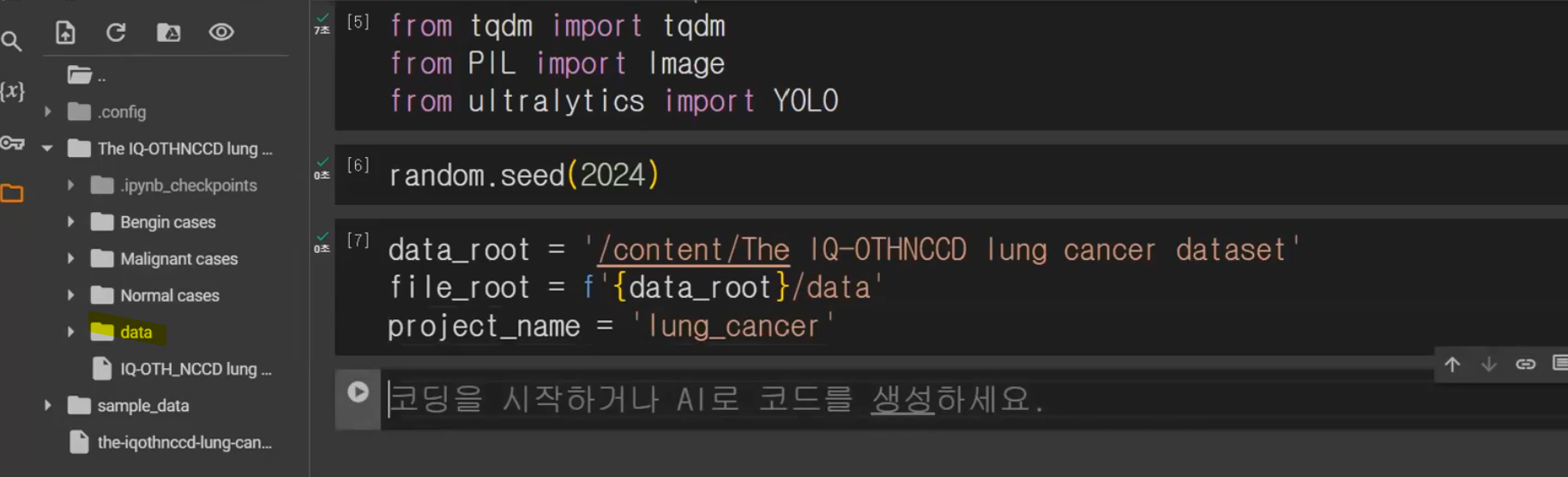
data_root = '/content/The IQ-OTHNCCD lung cancer dataset'
file_root = f'{data_root}/data'
project_name = 'lung_cancer'
data_root를 준비해두고 file_root와 project_name을 lang_cancer로 잡아주었습니다.
그냥 파일로하면 데이터가 잘 섞이지 않기때문에 train, valid, test로 나누어야겠습니다.
그래서 정리할 디렉토리를 정리해두도록 하겠습니다.
저는 코랩으로 진행했고 data를 파일을 하나 만들었습니다.(임의)
데이터 경로 지정 ↓
train_file_root = f'{data_root}/{project_name}'
train_root = f'{data_root}/{project_name}/train'
valid_root = f'{data_root}/{project_name}/valid'
test_root = f'{data_root}/{project_name}/test'
이렇게 되면 ex) train_root 는 data_root에 pjname에 train 에 됩니다.
이런식으로 train, valid, test로 폴더정의를 해주었습니다.
Bengin cases,Malignant cases,Normal cases디렉토리를 data 디렉토리 안으로 넣어주었습니다.
그래서 수동으로 끌어다 넣으면 경로가 완성이됩니다.
cls_list = os.listdir(file_root)
cls_list
이후에 for문으로 폴더를 만들어줍니다.
for folder in [train_root, valid_root, test_root]:
if not os.path.exists(folder):
os.makedirs(folder)
for cls in cls_list:
cls_folder = f'{folder}/{cls}'
if not os.path.exists(cls_folder):
os.makedirs(cls_folder)
train, valid, test를 가지고 가면서 folder에 넣고 os.path.exits(folder)에 존재하지 않으면
makedirs(폴더생성)을 하라고 하는것입니다.
그다음 cls_list(아까 위에서 만들었던 3개의 클래스를 뜻함) cls가 있으면 cls_folder를 가르키게 만들고
없으면 또 만들어달라고 합니다.

이렇게 파일로 잘 분리가 된것을 확인할 수 있습니다.
for cls in cls_list:
file_list = os.listdir(f'{file_root}/{cls}')
random.shuffle(file_list)
test_ratio= 0.1 #10%
num_file = len(file_list)
# print(num_file)
test_list = file_list[:int(num_file*test_ratio)]
valid_list = file_list[int(num_file*test_ratio):int(num_file*test_ratio)*2] #20%
train_list = file_list[int(num_file*test_ratio)*2:]
#파일들을 폴더에 맞게 넣기
for i in test_list:
shutil.copyfile(f'{file_root}/{cls}/{i}',f'{test_root}/{cls}/{i}')
for i in valid_list:
shutil.copyfile(f'{file_root}/{cls}/{i}',f'{valid_root}/{cls}/{i}')
for i in train_list:
shutil.copyfile(f'{file_root}/{cls}/{i}',f'{train_root}/{cls}/{i}')
여기서 섞은다음에 test_ratio는 10%만 사용할것이라고 합니다.
전체 파일리스트의 개수(416, 120,561)를 각각 cls list를 돌면서 각각 데이터를 넣어준것입니다.
그중의 10%를 파일리스트를 num*test_ratio를 하면 슬라이싱해서 10%만 곱해준것이됩니다.
마찬가지로 valid_list, test까지 ratio를 지정해줍니다.
각 list를 copyfile해서 옮겨주도록 해습니다.
새로고침해서 보면 각 파일에 데이터가 들어가있는것을 확인할 수 있습니다.
현재는 데이터가 섞여서 들어갔겠죠?
test_file_list = glob.glob(f'{test_root}/*/*')
random.shuffle(test_file_list)
#시각화
plt.figure(figsize=(20, 10))
for i in range(10):
test_img_path = os.path.join(test_root, test_file_list[i])
ori_img = Image.open(test_img_path).convert('RGB')
plt.subplot(2, 5, (i+1))
# /content/The IQ-OTHNCCD lung cancer dataset/lung_cancer/test/Bengin cases/Bengin case (10).jpg
plt.title(test_file_list[i].split('/')[-2])
plt.imshow(ori_img)
test_root에있는 모든 파일들을 다 가지고와서 test_file_list에 넣어줍니다.
그 부분또한 셔플을 넣어주고
파일이 어떻게 들어가있는지 확인해보기 위해서 plt로 시각화를 해보겠습니다.
각img들을 모두 연결해주어서 IMG를 RGB로 연결해주어서 보여주겠습니다.
그다음 -2가지 잘라주는이유는 경로를보면 슬러시를 기준으로 잘라내니까 CLASS이름이 되겠죠?
그 부분을 Title로 삼게됩니다!
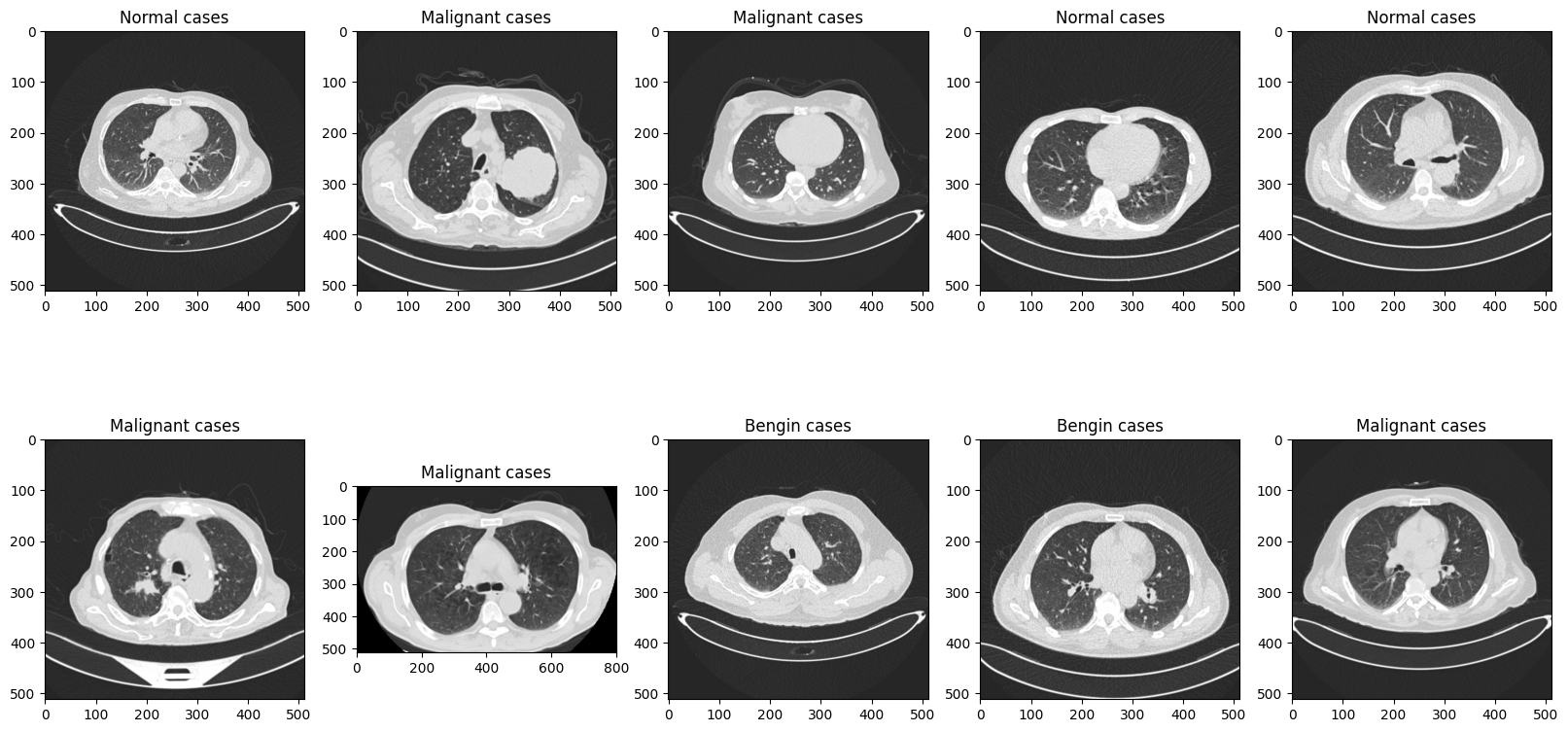
사실 비의료진으로서 normal과 begening이 어떤 차이인지는 잘 모르겠더라구요.
vision쪽은 정말 무궁무진하게 사용되는 분야이기때문에 다양한 도메인 지식을 접하고 학습할 수 있는 기회가 많이 생긴다고 하시는데 그 부분도 나에게는 굉장히 큰 cv의 매력으로 다가오게된다.
여기까지 해서 다시 변수로 넣어주는것이 좀 더 깔끔하고 편리할것같아서
변수로 정리해두겠습니다
project_root = '/content/The IQ-OTHNCCD lung cancer dataset/lung_cancer'🗒️ YAML 파일 만들기
yaml 이란?
yolo에 집어넣을 경로, 설정을 저장하는 파일입니다.
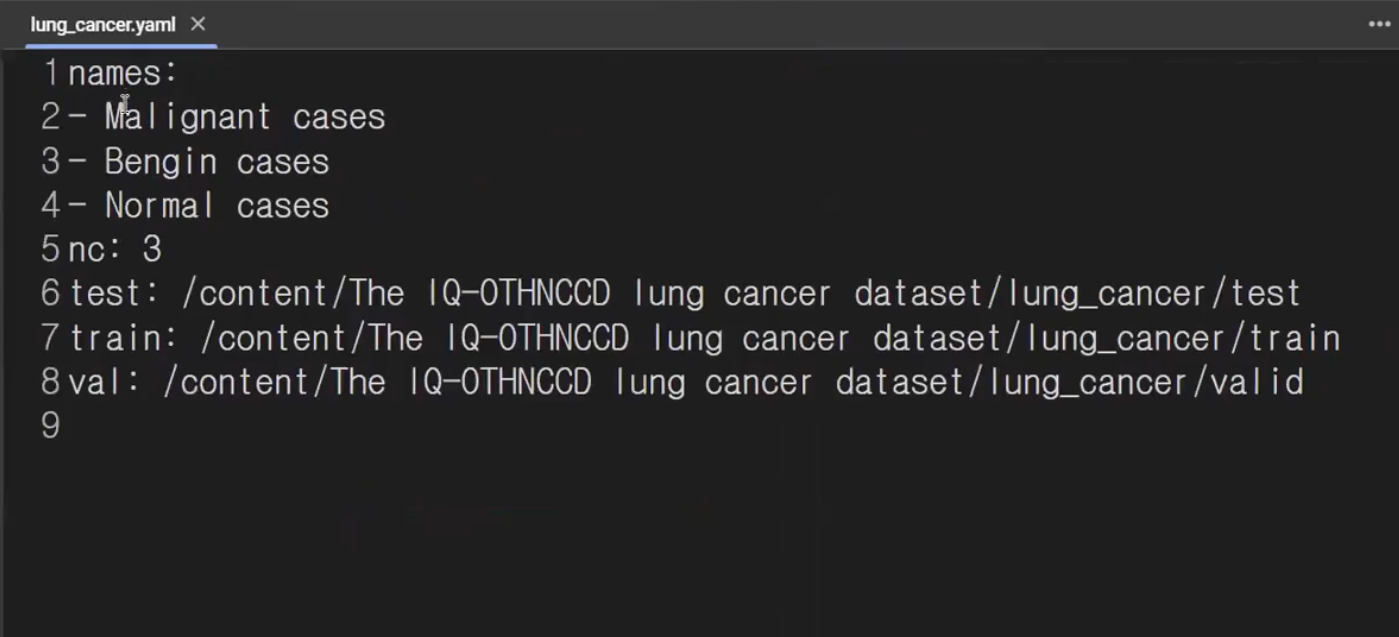
data= dict()
data['train']= train_root
data['val']=valid_root
data['test']=test_root
data['nc']= len(cls_list) #3
data['names'] = cls_list
#이렇게 정리한 딕셔너리를 파일로 저장하기
with open(f'{project_root}/lung_cancer.yaml', 'w') as f:
yaml.dump(data, f)
nc는 class의 개수입니다 #3개가 될것임
저렇게 정리한 딕셔너리를 파일로 저장하면 yaml파일로 만들어집니다.
🗒️ 버전 체크, 경로지정 하기
#현재 어떤 버전이 설치되어있는지 확인하는 방법
ultralytics.checks()
#경로지정
%cd /content/The IQ-OTHNCCD lung cancer dataset/lung_cancer
지금은 yolo를 깔지않았기댸문에 그냥 github에서 가져오는 방식으로 진행하겠씁니다.
#model = YOLO('내가 사용할 버전')
model = YOLO('yolov8s-cls.pt')
yolo를 실행하는 방법은
1. 변수를 만들어준다.
2. 파라미터를 넣어줍니다.
- patience : 조기종료(얼리스타핑) : 모델 성능이 더이상 개선되지않을때 조기종료를 수행하기 위한 기준을 설정함
예를들어 patience =30을넣으면 성능이 개선되지않는 에폭30번 이상 지속되면 조기종료합니다.
result = model.train(data=f'{data_root}/{project_name}', epochs=50, batch=8, device=0, name='lung_cancer_s', patience=30)
에포크 50번은 모두 돌아갔고 결과는 폴더에 저장이 되는것을 확인할 수 있습니다.
result_folder에 저장해줬습니다. 폴더 이름은 윗줄 name에서 따로 지정해주었어요.
result_folder= f'{project_root}/runs/classify/lung_cancer_s'🗒️ 예측모델
model = YOLO(f'{result_folder}/weights/best.pt')
예측모델은 YOLO를 가져올것이고 weights에 best.pt로 진행할것입니다.
test데이터를 가지고 검증을 하면서 얼마나 성능이 나오는지 확인하겠습니다.
그 결과를 메트릭스 라는 변수로 받습니다.
metrics = model.val(split='test')
여기서 top1 accuracy는 모델이 예측한 결과 중에서 가장 높은 확률로 예측한 값이 실제 정답과 일치하는 경우의 비율입니다.
하나를 뽑아서 정답인지 아닌지 확인하는경우에 사용합니다.
top5 acuuracy는 모델이 예측한 상위 5개의 결과 중 하나라도 정답을 포함할 경우의 비율을 나타냅니다.
print('top1 accuracy: ',metrics.top1)
print('top5 accuracy: ',metrics.top5)
위 코드의 print를 찍어보니
top1 accuracy: 0.9908257126808167
top5 accuracy: 1.0
가 나왔습니다.
🗒️ img예측 시각화 해보기
IMG_SIZE = (512, 512)
test_data_transform = transforms.Compose([
transforms.Resize(IMG_SIZE),
transforms.ToTensor()
])
이미지 데이터 전처리를 위한 변환(transform)작업을 합니다.
512,512사이즈 그리고 tensor형으로 변경해줍니다.
img = Image.open(test_file_list[0]).convert('RGB')
img_src = test_data_transform(img)
print(img_src.shape)
x_tensor = img_src.unsqueeze(0)
print(x_tensor.shape)
이미지 파일을열고 지정된 전처리를 거친 img를 Pytorch 텐서로 변환해서 모델입력으로 준비를 해줍니다.
그렇게 되면
torch Size가 ([3, 512, 512])
torch Size가 ([1,3, 512, 512])
- 이미지 열기 및 변환:
- Image.open(test_file_list[0]): test_file_list의 첫 번째 이미지 파일 경로를 사용하여 이미지를 엽니다.
- .convert('RGB'): 이미지를 RGB 모드로 변환합니다. 이는 이미지가 3채널(RGB) 색상 모드를 갖도록 보장합니다.
- 이미지 전처리:
- test_data_transform(img): 앞서 설정한 test_data_transform 변환을 적용하여 이미지를 전처리합니다. 이 변환은 이미지의 크기를 조정하고 텐서로 변환합니다. 결과는 PyTorch 텐서입니다.
- 전처리된 이미지의 형태 출력:
- img_src.shape: 전처리된 이미지 텐서의 형태를 출력합니다. 일반적으로 (채널 수, 높이, 너비) 형태를 가집니다. 예를 들어, torch.Size([3, 512, 512])는 3개의 채널(정상적으로 RGB 이미지)과 512x512 픽셀 크기의 이미지 텐서를 나타냅니다.
- 배치 차원 추가:
- img_src.unsqueeze(0): 텐서에 배치 차원(batch dimension)을 추가합니다. unsqueeze(0)은 텐서의 첫 번째 차원에 새로운 차원을 추가하여 (1, 채널 수, 높이, 너비) 형태를 만듭니다. 이는 모델이 배치 단위로 데이터를 처리할 때 유용합니다.
- 배치 차원이 추가된 텐서의 형태 출력:
- x_tensor.shape: 배치 차원이 추가된 텐서의 형태를 출력합니다. 예를 들어, torch.Size([1, 3, 512, 512])는 배치 크기가 1인 텐서로, 1개의 이미지(3 채널, 512x512 크기)가 포함된 것을 나타냅니다.
test_file_list[0]
/content/The IQ-OTHNCCD lung cancer dataset/lung_cancer/test/Normal cases/Normal case (132).jpg🗒️ 모델에 이미지를 입력해서 예측 결과를 얻기
result = model(x_tensor)[0]model(x_tensor) 모델을 호출해서 예측을 수행합니다.
x_tensor는 모뎁입력으로 사용되는 이미지 텐서고 모델의 forward메서드가 호출되어 예측결과를 반환합니다.
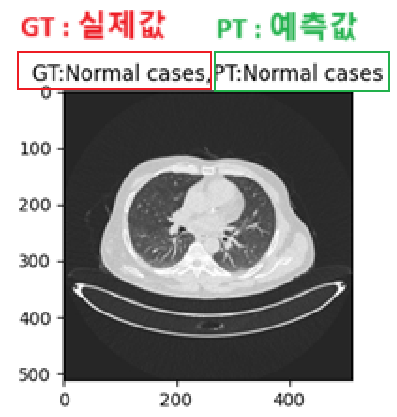
🗒️result가지고 시각화를 했고 이부분은 실제값입니다.
gt = test_file_list[0].split('/')[-2]
gtNormal cases로 확인됩니다.
🗒️예측
#예측
print(torch.argmax(result.probs.data).item())
print(model.names)
pt = model.names[torch.argmax(result.probs.data).item()]
pt2
{0: 'Bengin cases', 1: 'Malignant cases', 2: 'Normal cases'}
Normal cases🗒️시각화
plt.figure(figsize=(3,3))
plt.title(f'GT:{gt},PT:{pt}')
plt.imshow(np.array(img))
plt.show()
plt.figure(figsize=(20,5))
for idx in range(5):
img = Image.open(test_file_list[idx]).convert('RGB')
img_src = test_data_transform(img)
x_tensor = img_src.unsqueeze(0)
result = model.predict(x_tensor)[0]
gt = test_file_list[0].split('/')[-2]
pt = model.names[torch.argmax(result.probs.data).item()]
plt.subplot(1, 5, (idx+1))
plt.title(f'GT:{gt}, Predict:{pt}')
plt.imshow(img)
plt.show()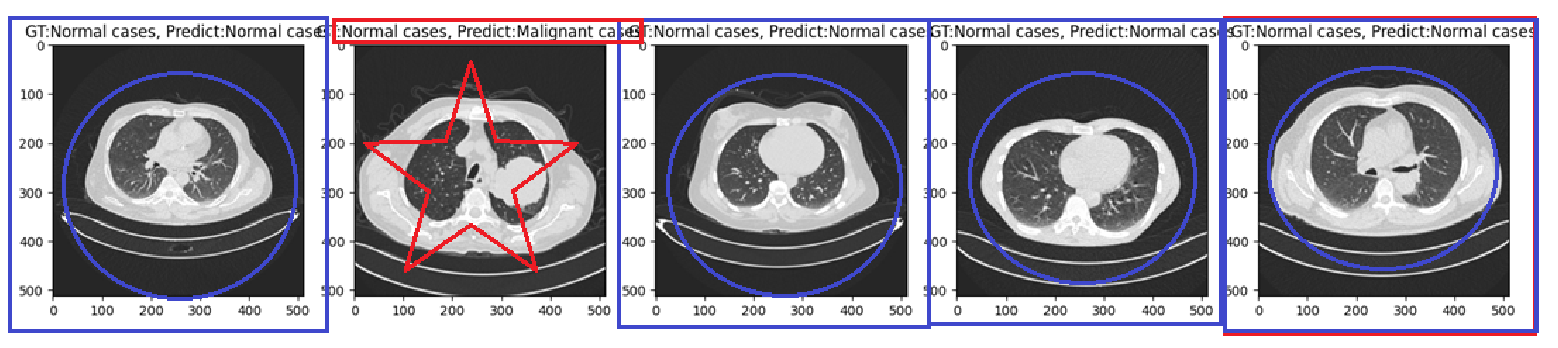
5개중에 1개빼고는 모두 잘 맞춘것을 확인할 수 있습니다.
여기서 중간에 PT가 아니라 Predict로 나온이유는 위에 title에서 predict로 썼기때문입니다.
pt로 변경해준다면 pt가 됩니다.
'AI > 컴퓨터 비전' 카테고리의 다른 글
| VGG19를 이용한 표면 균열(crack) 감지해보기 | 2개분류 (0) | 2025.03.09 |
|---|---|
| Computer Vision_개념을 다시한번 잡기 위한 글 (1) | 2025.03.06 |
| [DL] 모폴로지 처리, 도형이미지 구분하기 (1) | 2024.10.08 |
| [DL] 엣지검출(Canny) , 필터캠 (0) | 2024.10.07 |
| [DL] 투시변환(persective), 명함을 이용한 사진 활용 (5) | 2024.10.06 |



